Nos tutelles



Émergence et Adaptation des virus du riz
Objectifs
Notre équipe est focalisée sur l’identification des facteurs d’émergence et d’adaptation des virus du riz. Notre objectif principal est de contribuer à la définition et l’optimisation de stratégies de contrôle adaptées et durables face aux maladies virales.
En effet, les virus constituent une contrainte biotique majeure pour l’agriculture. La lutte contre ces agents pathogènes repose essentiellement sur la sélection de variétés résistantes. Cependant, la vitesse d’adaptation des virus peut parfois être très rapide et conduire à un effondrement des résistances variétales. Les facteurs impliqués dans l’émergence de génotypes virulents peuvent être à la fois intrinsèques (diversité génétique virale) et extrinsèques (paramètres liés à l’hôte, au vecteur ou à l’environnement).
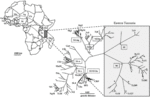
Nos travaux de recherche sont essentiellement centrés sur le virus de la panachure jaune du riz (Rice yellow mottle virus , RYMV) qui peut causer d’importantes pertes de récoltes chez la plupart des pays producteurs de riz en Afrique.
Des collaborations à long terme avec des chercheurs de plusieurs organismes de recherche africains permettent une connaissance détaillée de la diversité moléculaire et pathogénique du RYMV. L’histoire évolutive du virus est prise en compte à différentes échelles spatio-temporelles dans une démarche croisant approche expérimentale et comparative. Les capacités d’adaptation du virus sont explorées dans le contexte d’une agriculture en mutation et soumise aux changements climatiques.
Faits marquants
Emergence
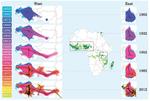
Suivi épidémiologique. Le virus continue d’être décrit dans de nouveaux pays rizicoles africains. Plant Disease 2012, 2013, 2014 , 2015. De nouvelles souches recombinantes ou non sont mises en évidence en particulier en Afrique de l’Est. Virus Res. 2015, Microbio. Res. Announc. 2017, 2018.
Diversité génétique et histoire évolutive du RYMV. Le virus a évolué sous forte pression de sélection, la pression de sélection a varié selon les lignées. L’hypothèse d’horloge moléculaire a été vérifiée. Virology 2009.
Rôle clé des étapes précoces de l’intensification de la riziculture dans l’émergence du RYMV en Afrique. En Afrique de l’Est, l’émergence virale a suivi l’extension et l’intensification. En Afrique de l’Ouest, l’histoire contrastée du riz a façonné les populations virales. . Current Opinion in Virology 2015.
Modélisation de la dynamique spatio-temporelle du RYMV. Le centre d’origine du virus se situe en Tanzanie, l’émergence sur riz a eu lieu il y a environ 200 ans. Après un évènement unique de transmission à longue distance d’Est en Ouest, les populations virales se sont maintenues de manière indépendante. Le rôle des facteurs environnementaux dans la dispersion du virus est en cours d’étude. . Virus Evolution 2015, 2018.
Adaptation
Résistance et Durabilité. Trois gènes impliqués dans la résistance élevée au RYMV ont été identifiés dans différentes accessions de riz africain cultivé (O. glaberrima). Plant Journal 2006, Frontiers in Plant Science 2016, Theoretical and Applied Genetics 2017. Cependant, la durabilité des résistances est variable, une carte de risques de contournement a été établie pour optimiser les stratégies de déploiement. Phytopathology 2018.
La capacité d’adaptation dépend de la souche virale. L’histoire évolutive passée du RYMV a une influence sur ses capacités d’adaptation actuelle. PLoS Pathogens 2007, Virology 2010 et PLoS Pathogens 2012.
Le cycle viral nécessite une interaction directe entre la VPg (viral protein genome-linked) et le facteur d’initiation de la traduction eIF(iso)4G1 du riz. La résistance médiée par les allèles du gène RYMV1implique des mutations dans eIF(iso)4G1 qui empêchent l’interaction, les mutations de virulence la restaurent. MPMI 2010.
Les mutations de virulence augmentent la valeur sélective des génotypes de RYMV dans les plantes résistantes. Certaines d’entre elles sont contre-sélectionnées dans les plantes sensibles. MPMI 2010.
Autres pathosystèmes étudiés:
Afrique/Amérique du Sud Asie du Sud-Est Afrique de l’Ouest
Plant Disease 2015. Virus Genes 2013.
Projets de recherche en cours
E-SPACE : a network for EpidemioSurveillance of PAthogens on Crops and their Environment
CRP RICE : CGIAR Research Project Rice, FP4 Global Rice Array
CRP GLDC : CGIAR Research Project Grain Legumes and Dryland Cereals, FP3 et FP4
LMI Patho-Bios : Observatoire des Agents phytopathogènes en Afrique de l’Ouest
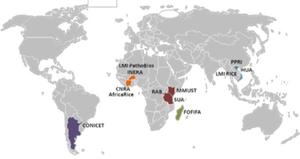
Principaux partenaires
Appui et Formation
Notre équipe participe au renforcement des capacités de chercheurs, techniciens et étudiants au Sud sous différentes formes : co-directions et co-encadrements de thèses, échanges scientifiques et accueils au laboratoire, formations théoriques et techniques ou ateliers in situ, appui à l’installation de laboratoires de diagnostic, missions de terrain et échantillonnages.
| Atelier de formation au PPRI (Vietnam) |
Soutenance de thèse à l’Université UFHB (Côte d’Ivoire) |
| Accueil d’étudiants et chercheurs à l’IRD Montpellier |
Installation de laboratoire à l’Université MMUST (Kenya) |
| Formation au LMI PathoBios (Burkina-Faso) |
Mission de terrain MMUST-IRD (Kenya) |
| Mission de terrain PPRI-IRD (Vietnam) |
Mission de terrain INERA-IRD (Burkina-Faso) |
Membres de l'équipe
- Hébrard Eugénie, Directrice de recherche IRD, Responsable d'équipe
- Aribi Jamel, technicien IRD
- Fargette Denis, Directeur de Recherche IRD
- Pinel-Galzi Agnès, Ingénieure IRD
- Poulicard Nils, Chargé de recherche IRD
IPME membre de l'I-Site