Nos tutelles



Nos projets de recherche
Sommaire
Nos activités de recherches sont en général financées et organisées sous la forme de projets multipartenariaux limités dans le temps et dont les objectifs sont définis au moment de l'appel d'offres. Voici nos principaux projets récents et en cours:
PIXIES: Rôle d'une nouvelle classe de petits ARNs du riz dans les maladies provoquées par Xanthomonas oryzae
Financement ANR 2015-2018. Partenaire: IPME Montpellier
Le projet PIXIES se propose d'étudier une stratégie de virulence originale des pathogènes bactérien qui s’appuierait sur l'extinction de l'immunité de l'hôte. Nous avons identifié plusieurs petits ARNs atypiques qui s'accumulent fortement dans la feuille de riz infectée par des souches virulentes de Xanthomonas oryzae . Ces petits ARNs induits par Xanthomonas (pARNix) possèdent des caractéristiques rappelant celles des petits ARNs régulateurs et pourraient cibler des gènes apparentés impliqués dans la signalisation de l'immunité végétale. Plusieurs approches expérimentales seront mises en œuvre afin de comprendre les facteurs bactériens et les voies de biosynthèse végétales nécessaires à la production des pARNix. La nature du mode d'action des pARNix à l'échelle moléculaire ainsi que l'identité de leurs cibles seront aussi précisés. Finalement, afin d'établir la pertinence biologique des pARNix, leur lien fonctionnel avec la mise en place de la maladie sera mis en évidence.
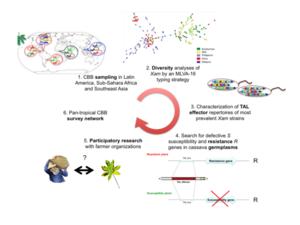
PAIX: PAthogen-Informed sustainable resistance of cassava against Xanthomonas
Cassava bacterial blight (CBB) is caused by the bacterial pathogen Xanthomonas axonopodis pv. manihotis (Xam ) which threatens cassava production worldwide. Yet no genetic resistance is available against CBB, and little is known about the diversity of Xam and how human practices influence itspopulation genetic structure. Our project aims at feeling these gaps by developing pathogen-informed innovative resistance strategies based on the diversity analysis of Xam populations in three contrasted cassava production areas. Search for resistance and loss-of-susceptibility cassava genes against Xam representative strains will guide the breeding of locally-adapted resistance into regional varieties.
PAPTiCROPs: Aptamer-based peptide interference with type III effectors for broad-spectrum and durable resistance of crop plants
Financement ANR 2016-2021.
Les bactéries phytopathogènes constituent un des obstacles majeurs à la production durable et eco-responsable de notre alimentation. La pathogènie de la majorité de ces bactéries, y compris Ralstonia solanacearum and Xanthomonas spp. dépend de la capacité à injecter à injecter les protéines effectrices de type 3 (T3E) dans la cellule végétale pour y manipuler la physiologie et l’immunité de l’hôte. Dans ce projet, nous voulons neutraliser ces bactéries phytopathogènes en inhibant l’activite de T3E conservés et importants pour la pathogènie de telle manière à limiter les possibilités de contournement de la résistance par le pathogène. Aussi, nous allons sélectioner, valider et utiliser des peptides aptamères de trois classes distinctes capables d’inhiber les T3E et/ou de les diriger vers des voies de dégradation de la cellule végétale. Cette interférence par peptides apatamères sera déployée chez la tomate et le riz qui sont hautement sensibles à R. solanacearum and X. oryzae .
SymEffectors: Système de secrétion de type 3 pour la symbiose fixatrice d'azote
Financement ANR 2016-2021.
Les légumineuses, de par leur capacité à s’associer symbiotiquement avec des bactéries fixatrices d'azote, appelées rhizobia , ont un impact écologique et agronomique majeur. Une avancée fondamentale dans notre compréhension de cette interaction symbiotique a été la découverte, au début des années 1990, que celle-ci repose sur un échange de molécules signal diffusibles entre les deux partenaires. Les facteurs Nod (FN) ont été reconnus comme un signal bactérien essentiel pour déclencher le programme de nodulation chez la plante hôte. Pendant longtemps, ce signal clé a été supposé être conservé chez tous les rhizobia capables de noduler. Cependant, en 2007, nous avons remis en question ce dogme en démontrant que des souches de Bradyrhizobium photosynthétiques nodulent certaines espèces d’Aeschynomene en l'absence de synthèse des FNs. Les signaux bactériens permettant l’établissement de cette symbiose restent à découvrir. Il a été proposé que ce processus symbiotique alternatif représente l'état ancestral de la symbiose rhizobium-légumineuse. Plus récemment, nous avons démontré que des bradyrhizobia non-photosynthétiques, grâce à un système de sécrétion de type 3 (T3SS), peuvent également activer la nodulation chez les Aeschynomene indépendamment des FNs. Etant donné que cette machinerie de sécrétion est absente chez la plupart des souches de bradyrhizobia photosynthétiques, nous concluons qu’il existe au moins deux voies de signalisation indépendantes des FNs, une qui nécessite le T3SS et l’autre pas. Le complexe T3SS, initialement identifié chez les bactéries pathogènes de plantes et d’animaux, permet l’injection de T3SS effecteurs à l'intérieur des cellules hôtes où ils suppriment les réactions de défense et favorisent l'infection. Bien qu'il soit connu depuis plus d'une décennie que les rhizobia peuvent utiliser le T3SS pour moduler le système immunitaire de la plante lors de la nodulation, notre travail indique pour la première fois que des T3SS effecteurs peuvent aussi activer directement la nodulation en contournant la reconnaissance des FNs.
Notre objectif principal dans le projet SymEffectors est de décortiquer les mécanismes moléculaires de ce processus symbiotique alternatif, T3SS-dépendant. Notre consortium, impliquant des spécialistes de la symbiose et pathogenèse végétale, combinera des approches omiques, de la génétique bactérienne et végétale ainsi que des techniques de biologie cellulaire pour disséquer au niveau moléculaire cette nouvelle voie symbiotique. Nous allons nous focaliser sur le questionnement scientifique suivant: Quels sont les T3SS effecteur(s) qui déclenchent l’organogenèse nodulaire et l'infection? Quelles sont les voies de signalisation de la plante qui sont activées lors de la mise en place de ce processus symbiotique? Quel est le degré de conservation entre les 2 voies de signalisation symbiotiques indépendantes des FNs? Quelles sont les cibles chez la plante de ces T3SS effecteur(s)? Les connaissances sur ce programme de nodulation ancestral devraient être très importantes pour mieux exploiter les interactions symbiotiques rhizobium-légumineuses déjà existantes dans la nature mais aussi pour le développement de nouvelles stratégies de transfert de la symbiose fixatrice d'azote aux céréales.
EuroXanth: Integrating science on Xanthomonadaceae for integrated plant disease management in Europe
Financement COST 2016-2020.
Bacteria of the family Xanthomonadaceae , including species of Xanthomonas and Xylella fastidiosa , belong to the most devastating plant pathogens continually challenging food security. Many of the pathogens are listed as quarantine organisms in the EU and their study is of uttermost importance. The concerned pathogens infect all kinds of crop plants, such as cereals, forage crops for ruminant feed, vegetables, fruits, shrubs and trees.
This COST Action will bring together some of the brightest and best minds to join in an interdisciplinary network to develop strategies for sustainably protecting plants and significantly reducing yield losses. Specifically, this initiative will address several key aspects of the pathogen-vector-host interactions from the cellular to the population level. A better insight into population structures and virulence mechanisms of the pathogens, together with the exploration of the molecular mechanisms underlying disease resistance to the pathogen, will enable development of durably resistant plant cultivars and exploitation of bio-control schemes tailored to population and pathogen.
This Action will generate a platform that gathers experts from different disciplines, such as molecular diagnostics, molecular host-microbe interactions, plant resistance breeding, and applied microbiology. Joining their efforts will help to develop and implement effective plant protection schemes, be it via resistant crop cultivars or via other control mechanisms. This goal will be achieved by mobilizing and training scientists from major European institutions, regulatory bodies and commercial companies working on the various aspects of this complex of problems.
MUSEOVIR: Étude des ADN anciens de pathogènes présents dans les herbariums
Financements:
CRESI CIRAD: exploration, mise au point technique, levée des verrous méthodologiques
Fondation Agropolis: exploration, création d’un réseau de scientifiques et curateurs d’herbiers
E-Space (Fondation Agropolis): cadre global, lien avec questions d’émergence…
Partenaires: IPME Montpellier, BGPI Montpellier, PVBMT La Réunion, ISYEB Paris (Muséum d'Histoire Naturelle)

Plusieurs projets imbriqués sont consacrés à la collecte et l’analyse d’acides nucléiques dans les échantillons d’herbiers anciens. L’objectif global est la reconstruction des routes d’invasion et de l’histoire évolutive des agents pathogènes. À l’approche traditionnelle de génétique des populations habituellement appliquée à des échantillonnages modernes nous souhaitons ajouter des données issues d’échantillons d’herbiers pouvant dater de plus de 200 ans. Cette approche peut permettre d’identifier la présence d’un pathogène sur une plante donnée à une date et dans un lieu donné. L’étude des acides nucléiques microbiens présent dans ces échantillons permettra de décrire et dater le plus récent ancêtre commun des souches modernes, identifier les paramètres clés de l’émergence et/ou de la spécialisation d’hôte, évaluer la vitesse d’accumulation des mutations dans les génomes.
CROPTAL: Ingénierie de la résistance des plantes cultivées aux pathogènes basée sur le TALome

Financement ANR 2015-2018. Partenaires: LIPM Toulouse, PVBMT La Réunion, IRHS Angers, IPME Montpellier
En l'absence de méthodes chimiques efficaces et non toxiques, la sélection de variétés génétiquement résistantes reste la stratégie la plus prometteuse pour lutter durablement contre les bactéries phytopathogènes. Néanmoins, cette approche nécessite des sauts conceptuels dans les modes de sélection de variétés d'intérêt agronomique résistantes. Le projet CROpTAL cherche à identifier des cibles de sensibilité conservées dans des familles d'intérêt agronomique majeur (céréales, agrumes, brassicacées, légumineuses). Les cibles de sensibilité seront identifiées grâce aux effecteurs TALs (Transcription Activator-Like) de Xanthomonas qui activent l’expression de gènes de sensibilité chez la plante afin de promouvoir la maladie. Des variants inactifs de ces cibles de sensibilité conservées au sein d'une espèce pourront alors être utilisés dans des programmes de sélection assistée par marqueurs pour aider à l'élaboration de variétés durablement résistantes aux agents pathogènes.
MIC-CERES: Microbial eco-compatible strategies for improving wheat quality traits and rhizospheric soil sustainability
Financement Agropolis et Cariplo 2014-2017. Partenaires: IPME Montpellier (équipe ABIP et GTIPP), University of Insubria (Italy), University of Torino (Italy), CRA-GPG, Genomics Research Center (Italy), UMR Microbial Ecology (Villeurbanne, France), Common laboratory of Microbiology (LCM, Dakar, Senegal), Institute of Agronomic Research and Development (IRAD, Yaounde, Cameroon)
http://umr-lstm.cirad.fr/principaux-projets/mic-ceres
XANTHOMIX: Etude comparative des génomes et des transcriptomes de Xanthomonas phytopathogènes
Financement ANR 2011-2014. Partenaires: LIPM Toulouse, PVBMT La Réunion, IRHS Angers, IPME Montpellier
XANTHRACING : Nouvelles méthodes de traçage appliquées aux Xanthomonas pathogènes du riz et des citrus
Financement ANR 2011-2014. Partenaires: IGM Orsay (CNRS), PVBMT La Réunion, IPME Montpellier
Des régions répétées à évolution rapide comme nouveaux outils de typage moléculaire pour la surveillance épidémiologique des Xanthomonas phytopathogènes
L'objectif principal de notre projet était de développer un nouveau système de typage moléculaire simple, rapide, sensible et robuste qui soit utile pour le suivi de X. oryzae pv. oryzae et X. citri pv. citri . L'originalité et la nouveauté du projet résidaient dans l'identification, la caractérisation et l'exploitation des régions répétées qui évoluent plus rapidement que le reste du génome, permettant ainsi leur utilisation pour la surveillance épidémiologique. Plus précisément, nous avons exploré deux types de locus:
- les loci VNTR (nombre variable de répétitions en tandem, également connu sous le nom de satellites),
- le locus CRISPR (groupe de courtes répétitions palindromiques régulièrement espacées).
Sur la base de données de génomes, nous avons évalué 163 loci candidats identifiés comme satellites et développé des outils de typage VNTR pour les deux agents pathogènes. Des schémas de typage multiplex, permettant l'analyse simultanée de quatre loci satellite par souche (16 loci de Xoo et 31 loci pour Xci), ont été appliquées à plus de 200 souches de chaque espèce. Ce nouveau système de typage/traçage semble être hautement reproductible et universellement applicable et peut être réalisé à faible coût dans différents laboratoires et environnements utilisant un équipement normal.

Illustration des approches de typage moléculaire pour Xci , causant le chancre bactérien (en haut à gauche), et Xoo , causant le dépérissement foliaire (en bas à droite). Les 2 pathogènes ont été étudiés pour leurs satellites, comme illustré par l’électrophorégramme montrant le polymorphisme de taille des amplicons PCR de différentes souches (en bas à gauche), et pour les loci CRISPR qui consistent en des répétitions identiques espacées par des séquences uniques (en haut à droite). © R Koebnik, O Pruvost
Le projet XANTHRACING a généré onze manuscrits publiés dans des revues internationales évaluées par des pairs. Les outils et principes de ce travail ont également été appliqués à d'autres Xanthomonas , comme X. axonopodis pv. manihotis pathogène du manioc. En outre, le projet a généré des dizaines de séquences préliminaires du génome de Xoo et Xci qui ont permis de comparer la résolution du typage VNTR avec le typage CRISPR.
Pour faciliter les analyses de données et permettre des comparaisons inter-laboratoires, nous avons mis en place une ressource de typage moléculaire basée sur Internet pour les bactéries pathogènes des plantes, et qui rend nos données accessibles au public sous la forme de la base de données MLVABANK (http://www.biopred.net/MLVA/). Cette base de donnés sert la communauté scientifique internationale et fournit aux organismes de surveillance des outils de surveillance épidémiologique des maladies bactériennes.
Le projet XANTHRACING était un projet de recherche fondamental coordonné par Ralf Koebnik, UMR RPB Montpellier. Il a associé les équipes dirigées par Olivier Pruvost (UMR 53 PVBMT Saint-Pierre La Réunion) et Christine Pourcel (UMR 8621 IGM Paris-Sud Orsay).
Permanents de l'équipe
- Boris Szurek Directeur de Recherche IRD, Responsable d'équipe
- Florence Auguy Ingénieure IRD
- Sébastien Cunnac Chargé de Recherche IRD
- Lionel Gagnevin Chercheur CIRAD,
- Mathilde Hutin Chargée de Recherche IRD
- Ralf Koebnik Directeur de Recherche IRD
- Valérie Verdier Directrice de Recherche IRD
- Emmanuel Wicker Chercheur CIRAD
Pour plus d'info
IPME membre de l'I-Site