Nos tutelles



Génomique & Transcriptomique des Interactions Plantes-Procaryotes
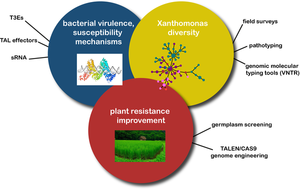
Les projets de l’équipe GTIPP sont principalement centrés sur les interactions entre les bactéries du genre Xanthomonas et ses hôtes, notamment chez le riz, le manioc et les agrumes. Ils s’articulent autour de trois axes majeurs:
- l’étude des facteurs impliqués dans le pouvoir pathogène des Xanthomonas et de la résistance chez l’hôte (au niveau de la cellule, des tissus et de l’organisme),
- des outils de typage génétique pour la compréhension de la diversité et de l’évolution des agents pathogènes (niveau des populations).
- le développement et l'application de nouveaux outils pour la modification ciblée du génome et le transfert des connaissances et des technologies vers l'Afrique subsaharienne.
Les études sur les bactéries pathogènes se caractérisent par une approche pluridisciplinaire et s’appuient sur un partenariat bilatéral et multilatéral diversifié.
Modèles biologiques étudiés
Les Xanthomonas sont un genre bactérien contenant une 20aine d'espèces s'attaquant à plus de 140 plantes. Chaque espèce est divisée en pathovars, reflétant une spécialisation d'hôte et de symptomatologie. Ceci nous permet de travailler sur des modèles biologiques complémentaires (systémiques vs. parenchymateux, hôte pérenne vs. hôte annuel, maladie émergente vs. maladie endémique…) afin de déchiffrer les mécanismes de la spécialisation d'hôte et de tissus ainsi que les facteurs conduisant à l'émergence.
Thèmes de recherches
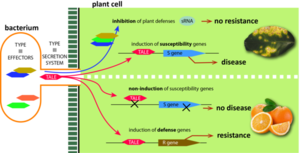
Les Xanthomonas utilisent un mécanisme original pour attaquer les plantes et causer les symptomes: le système de sécrétion de type 3 (SST3) et les effecteurs bactériens qu'il injecte à l'intérieur de la cellule végétale. Dans le cas où la plante est sensible ces effecteurs de type 3 vont inhiber les réactions de défense et manipuler le métabolisme de la cellule végétale au bénéfice de la bactérie. En particulier, les effecteurs TAL (TALE) activateurs transcriptionnels bactériens modulent l'expression de gènes cibles de la plante qui sont appelés gènes de susceptibilité. Lorsque la plante est résistante c'est qu'elle est capable de détecter la présence de ces effecteurs et déclencher la résistance précoce.
- Épidémiologie moléculaire, génétique des populations, pathotypage, diagnostic (Xvm, Xoo, Xoc, Xam, Xt )
- Diversité & évolution des SST3 & ET3 (Xac, Xoo, Xoc, Xt )
- Diversité & évolution des effecteurs TAL (Xac, Xoo, Xoc, Xt, Xam )
- Fonction & cibles des effecteurs TAL (Xac, Xoo, Xoc, Xt, Xam )
- Rôle des sRNA de riz dans la susceptibilité (Xoo , Xoc )
- Ingéniérie de la résistance : criblage de germplasm, édition de génome (riz, manioc)
Projets de recherche
- PIXIES: Rôle d'une nouvelle classe de petits ARNs du riz dans les maladies provoquées par Xanthomonas oryzae
- PAIX: PAthogen-Informed sustainable resistance of cassava against Xanthomonas
- PAPTiCROPs: Aptamer-based peptide interference with type III effectors for broad-spectrum and durable resistance of crop plants
- SymEffectors: Système de secrétion de type 3 pour la symbiose fixatrice d'azote
- EuroXanth: Integrating science on Xanthomonadaceae for integrated plant disease management in Europe
- MUSEOVIR: Étude des ADN anciens de pathogènes présents dans les herbariums
- CROPTAL: Ingénierie de la résistance des plantes cultivées aux pathogènes basée sur le TALome
- MIC-CERES: Microbial eco-compatible strategies for improving wheat quality traits and rhizospheric soil sustainability
- XANTHOMIX: Etude comparative des génomes et des transcriptomes de Xanthomonas phytopathogènes
- XANTHRACING : Nouvelles méthodes de traçage appliquées aux Xanthomonas pathogènes du riz et des citrus
Résultats marquants
-
Participation aux grands projets de séquençage des Xanthomonas
(consortium FNX): plusieurs centaines de génomes, des outils indispensables pour nos approches
(Gordon et al. 2015, BMC Genomics PMC4690215; Jacobs et al. 2015, Front Plant Sci PMC4468381) -
Développement d’outils de typage MLVA et application à l’étude des populations et suivi épidémique
(Poulin et al. 2015, Appl Environ Microbiol PMC4277570) -
Mise en place d’outils WEB de classification et annotation automatique des TALE, de prédiction des cibles végétales
(Perez-Quintero et al. 2015, Front Plant Sci PMC4522561; Perez-Quintero et al. 2013, PLoS One PMC3711819)- http://www.xanthomonas.org: des pages d'informations générales sur les Xanthomonas
- QUETAL: algoritme pour comparer et classifier les TALE selon leur séquence totale et la séquence RVD
- TALVEZ: algoritme pour prédire les cibles végétales des TALE à partir de la séquence des RVD et de la plante hôte
- Identification de petits ARN végétaux induits par la bactérie (ET3) pouvant supprimer l’immunité du riz
-
Identification d’un nouveau gène de résistance du riz à Xoo (xa41) par perte de sensibilité
(Hutin et al. 2015, Plant Journal Link) -
Caractérisation des transporteurs de sucre de type SWEET comme gènes de sensibilité majeurs du riz à Xoo
(Hutin et al. 2015, Front Plant Sci PMC4500901; Denancé et al. 2014, Plant Cell Physiol Link) - Identification d’UPTAL2, facteur de transcription de type ERF, comme nouveau gène de sensibilité spécifique des Xoo africaines
- Création de nouveaux allèles de résistance au BLB (Xoo ) par édition du génome de riz (Blanvillain-Baufumé et al. 2016, Plant Biotechnology Journal Link)
- Développement de stratégies d’analyse des cibles de TALE par création de TALE artificiels
Partenaires
- Membres du
réseau FNX:
- LIPM (INRA, CNRS, UPS), Toulouse
- UMR BGPI (INRA, CIRAD, SupAgro), Montpellier
- IRHS-EMERSYS (INRA, AgroCampus Ouest, U. Angers), Angers
- UMR PVBMT (CIRAD, U. Réunion), La Réunion
- LMI Patho-Bios (Burkina-Faso) ; JEAI CoANA (Mali)
- CIAT, IRRI, CRP RTB, CRP RICE, AfricaRice, Bioversity International, IITA, FERA…
- U. of Colorado, Cornell U. (USA), U. de los Andes, U. Nacional (Colombie), U. Catholique Louvain (Belgique), Leibniz Universität Hannover (Allemagne), U. Exeter (UK), Agriculture Genetics Institute (Vietnam)
Membres de l'équipe
Notre équipe est actuellement constituée de 6 chercheurs, 2 ingénieures, 2 chercheurs post-doctorants, 5 doctorants
Publications des membres de l'équipe en 2017
Publications des membres de l'équipe en 2016
Publications des membres de l'équipe en 2015
Publications des membres de l'équipe en 2014
Permanents de l'équipe
- Boris Szurek Directeur de Recherche IRD, Responsable d'équipe
- Florence Auguy Ingénieure IRD
- Sébastien Cunnac Chargé de Recherche IRD
- Lionel Gagnevin Chercheur CIRAD,
- Mathilde Hutin Chargée de Recherche IRD
- Ralf Koebnik Directeur de Recherche IRD
- Valérie Verdier Directrice de Recherche IRD
- Emmanuel Wicker Chercheur CIRAD
Pour plus d'info
IPME membre de l'I-Site